Vous êtes ici : FHU-SEPSISFRMISSIONS & PROJETSFeuille de routeWP3 : Plateforme "multi-omiques"
- Partager cette page :
- Version PDF
WP3 : Plateforme "multi-omiques"
Leader : Pr Stanislas Grassin Delyle - Co-leader : Pr Henri Jean Garchon
Partenaires : UVSQ/INSERM/CEA/AP-HP/Hôpital Foch
L’objectif de la FHU SEPSIS est l’obtention de tests rapides "au lit du malade" dont le développement repose sur l’identification de biomarqueurs d’intérêt grâce aux technologies les plus performantes, le traitement des données étant également un enjeu majeur du succès de ces approches. Au cours de la dernière décennie, les technologies "omiques" ont connu un essor majeur, et les approches "multi-omiques" intégrées allient les intérêts et les spécificités de la génomique, la transcriptomique et la métabolomique. La génomique et la transcriptomique permettent une analyse approfondie de la variation génétique ainsi que des multiples niveaux d'expression des gènes. La métabolomique est consacrée à l’étude des métabolites, molécules de petit poids moléculaire, essentiellement par spectrométrie de masse. Ces approches sont habituellement mises en œuvre dans les fluides biologiques ou les tissus, mais plusieurs centaines de métabolites à faible point d’ébullition, appelés composés organiques volatils, peuvent également être mesurés dans l’air expiré où ils sont aussi le reflet de l’activité métabolique de l’organisme et de la pathologie.
Plusieurs plateformes et laboratoires dans le périmètre de l’UVSQ / Université Paris-Saclay vont ainsi être impliquées dans les projets de recherche de la FHU. En premier lieu, les plateformes du Département de Biotechnologie de la Santé (UFR Simone Veil – Santé), et parmi elles les plateformes de génomique et de spectrométrie de masse, disposant des instruments et de l’expertise nécessaire à la mise en œuvre de l’ensemble des approches "omiques". La plateforme du CNRGH (CEA) sera également sollicitée pour le séquençage du génome entier. Ensuite, la plateforme Exhalomics® (Hôpital Foch / UVSQ) possède une gamme d’outils technologiques comme des nez électroniques et des spectromètres de masse dédiés à des projets de recherche clinique d’analyse de l’air expiré. Le traitement des données sera en partie réalisé par les plateformes concernées, mais également dans le cadre d’une collaboration avec l'équipe Sciences des Données pour le Phénotypage Moléculaire et la Médecine de Précision (SciDoPhenIA, Dr Thévenot, CEA, Département des technologies du médicament et des sciences de la vie), spécialisée dans le traitement, l'analyse et l'intégration des données de métabolomique et de volatolomique.
La force de la FHU sera de proposer à ces plateformes des échantillons de grandes cohortes de patients parfaitement caractérisés, en s’appuyant sur des cohortes préalablement constituées ainsi que sur l’essai clinique adaptatif du RHU RECORDS.
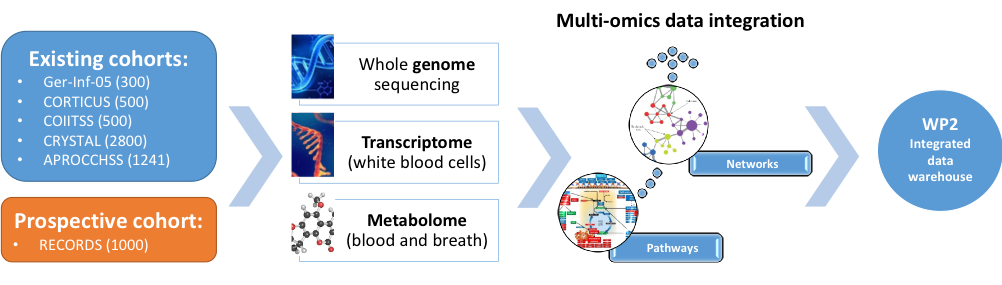
L’organisation générale du WP est représentée dans la figure ci-dessous. Les signatures "multi-omiques" intégrées seront établies à partir de cohortes existantes ou en cours de constitution, à l’aide des outils technologiques mis à disposition par un ensemble de plateformes et en utilisant des méthodes statistiques de pointe qui intègrent l'ensemble de données multidimensionnelles à haut niveau générées, grâce à l’expertise présente dans les plateformes existantes ou d’équipes participant au WP. Les signatures établies seront ensuite transmises au WP2 pour intégration avec l’échelon supérieur, intégrant l’ensemble des données démographiques, cliniques, biologiques, thérapeutiques et épidémiologiques des patients.
Objectifs et organisation du WP3